Científicos del IRB Barcelona, el IBMB-CSIC y el BSC descubren un nuevo método más inocuo para etiquetar proteínas
El Instituto de Investigación Biomédica de Barcelona (IRB Barcelona) y el Instituto de Biologia Molecular de Barcelona (IBMB-CSIC), ambos ubicados en el Parc Científic de Barcelona, en colaboración con el Barcelona Supercomputing Center (BSC) y la Universidad de Barcelona (UB) participan en un estudio que ofrece un nuevo método para hacer un seguimiento más limpio de proteínas dentro de las células.
El estudio realizado en las instalaciones del Parc Científic de Barcelona, sede de dos de los dos institutos que han participado, ha revelado un nuevo método para etiquetar proteínas que preserva funciones nativas y su estructura.
Publicado hoy en Nature Methods, la revista más prestigiosa para presentar resultados de desarrollo de métodos, propone el uso de epítopos (un tipo de antígenos) de proteínas de dos plantas, denominados inntags, como herramientas más inocuas y estables de etiquetado para estudiar las interacciones físicas y funcionales de proteínas.
Las proteínas y los péptidos de varios tamaños y formas se han utilizado desde los años 80 para etiquetar proteínas con muchos propósitos diferentes, que van desde la purificación a la detección microscópica basada en la fluorescencia en organismos completos. Sin embargo, en las estrategias de etiquetado utilizadas a día de hoy se corre el riesgo de que la función nativa de la proteína se anule o quede comprometida por las interacciones con la etiqueta.
Un estudio liderado por investigadores del Instituto de Biología Molecular de Barcelona del CSIC, del Programa Conjunto de Biología Computacional del Instituto de Investigación Biomédica (IRB Barcelona) y el Barcelona SuperComputing Center (BSC), y la Universidad de Barcelona, ha analizado la lista disponible de polipéptidos con estructura 3D conocida para identificar entre ellos los más adecuados para ser usados como etiquetas.
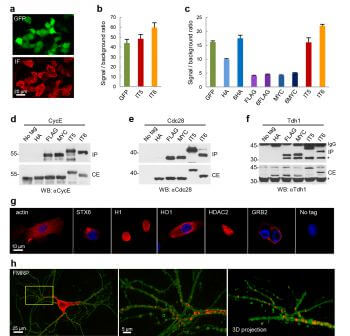
Los investigadores han seleccionado los dominios más pequeños de proteínas que aún muestran fuertes determinantes estructurales para actuar como antígenos, no generan problemas de solubilidad, no comprometen la salud de las células y no causan efectos detectables en función ni localización cuando se fusionan con otras proteínas diana.
Primero, los científicos usaron una serie de herramientas bioinformáticas para escanear el proteoma completo y seleccionar aquellas proteínas que podrían, en principio, tener buenas propiedades de marcado. Después de una examen manual de los resultados in silico, ensayaron en vivo 12 candidatos a etiqueta, encontrando para todos ellos excelentes propiedades. Los inntags mantienen su integridad, son estables, son solubles en extractos celulares, tienen movilidad de difusión y no causan perturbaciones funcionales importantes que sí causan otras etiquetas usadas habitualmente, como MYC, FLAG o HA.
La tecnología desarrollada abrirá nuevas posibilidades para los investigadores en biología celular ya que les proporcionará un método más ordenado e imparcial para realizar el seguimiento de proteínas dentro de la célula.




