Revelan cómo diseñar por ordenador proteínas artificiales para nuevas aplicaciones
Un equipo internacional de científicos liderados por la Universidad de Washington (UW) en Seattle ha descifrado las reglas clave que gobiernan cómo las proteínas forman estructuras con cavidades que son esenciales para muchas funciones clave de proteínas. El descubrimiento permite que los investigadores diseñen a medida proteínas que pueden imitar las acciones de proteínas naturales, así como diseñar proteínas desde cero, diferentes a cualquiera que se encuentre en la naturaleza, capaces de realizar funciones totalmente nuevas. Enrique Marcos, investigador de l'IRB Barcelona en el Parc Científic de Barcelona, és el primer autor del estudio que se publica hoy en Science.
Actualmente, los científicos que tratan de diseñar una nueva proteína para actuar sobre una molécula particular suelen reutilizar proteínas naturales, pero esta estrategia tiene varias limitaciones. El investigador Enrique Marcos, que actualmente trabaja en el laboratorio de Modesto Orozco del Instituto de Investigación Biomédica (IRB Barcelona), explica que «esta nueva estrategia nos permitirá depender menos de las proteínas naturales y diseñar proteínas con potencialidad para desarrollar nuevas pruebas de diagnóstico y tratamientos, o para propósitos biotecnológicos, como catalizar reacciones para conseguir procesos industriales más eficientes».
David Baker, director del Instituto de Diseño de Proteínas de la UW, qque dirigió la investigación, apunta que «este enfoque permitirá afinar el tamaño y la forma de estas cavidades o bolsillos para que las proteínas diseñadas a medida se pueden unir y actuar sobre una molécula específica, un proceso llamado unión de ligando”.
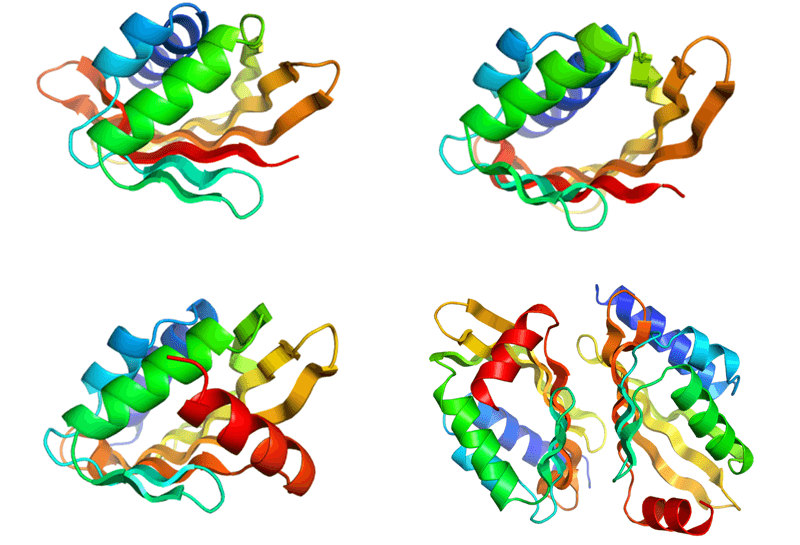
Las proteínas están formadas de cadenas de aminoácidos que se pliegan en una forma compacta que determina su función. El equipo estudió las estructuras que se forman cuando cadenas dentro de la misma proteina se alinean una junto a otra para crear unas estructuras laminares, llamadas hojas beta (hojas β). En muchas proteínas naturales, estas hojas se doblan para formar cavidades que se unen a las moléculas diana implicadas en muchos procesos celulares.
Para desarrollar la nueva estrategia, los científicos identificaron las propiedades clave en la secuencia y orientación de los aminoácidos que determinan cómo las hojas β se pliegan y se curvan. Una vez identificadas estas características, los investigadores muestran que es posible diseñar computacionalmente estructuras de proteínas y producirlas experimentalmente con alta precisión respecto del modelo computacional. Dichas proteínas forman cavidades con una variedad de formas y tamaños y además demuestran que son altamente estables y toleran altas temperaturas, lo que es esencial para la ingeniería de nuevas funciones.
• Artículo de referencia:
Enrique Marcos, Benjamin Basanta, Tamuka M. Chidyausiku, Yuefeng Tang,Gustav Oberdorfer, Gaohua Liu, G.V.T. Swapna, Rongjin Guan, Daniel-Adriano Silva, Jiayi Dou, Jose Henrique Pereira, Rong Xiao, Banumathi Sankaran, Peter H. Zwart, Gaetano T. Montelione, David Baker. «Principles for designing proteins with cavities formed by curved β-sheets«. Science (2017). Doi: 10.1126/science.aah7389




